Изменчивость ключевых генов, участвующих в формировании агрономических признаков, таких как качество зерна и характеристики хлебопечения, имеет решающее значение для селекции. Но сам процесс расшифровки последовательностей генов в больших коллекциях растений дорогой и длительный. С целью его оптимизации ученые из Всероссийского НИИ сельскохозяйственной биотехнологии и МФТИ апробировали новый метод расшифровки полноразмереных генов на примере шести, кодирующих глютенины — запасные белки зерновки. В результате ученые не только определили новый экономичный путь, но и обнаружили новый вариант (аллель) одного из генов глютенинов пшеницы. Исследование опубликовано в журнале Agronomy. Проект реализован при поддержке Минобрнауки РФ.
Расшифровка последовательности генов сельскохозяйственных растений дала мощный стимул для развития молекулярной селекции, позволяя путем ДНК-скрининга значительно быстрее находить и комбинировать ценные природные варианты генов, добиваясь лучших свойств и высокой урожайности. Этот метод значительно отличается от ГМО-технологии, которая добивается заданных свойств с помощью добавления новых генов.
«В природе существуют детерминанты — ключевые гены, состояние и последовательность которых определяют основные сельскохозяйственные признаки. Таких генов пока известно не так много, так как большинство признаков растений, например урожайность, контролируется большим числом генов и зависит от среды произрастания. При этом одни аллели — варианты генов — могут лучшим образом влиять на признак, чем другие. Это основа маркерной селекции, которая позволяет, не дожидаясь, пока признаки проявится в зерне, понять, каким будет его качество. В итоге мы экономим очень много времени, совершая оперативную отбраковку буквально на стадии проростка или подбирая наиболее удачные комбинации по генотипу для новых сортов. Например, с помощью расшифровки последовательности генов мы знаем, какой ген дает короткостебельные сорта пшеницы, а какое сочетание генов отвечает за хлебопекарные качества. Скрестив их, как кубик Рубика, мы отбираем генотип, который будет и короткостебельным, и с наилучшими качествами», — рассказывает Илья Киров, заведующий лабораторией маркерной и геномной селекции растений ВНИИ сельскохозяйственной биотехнологии и лабораторией системной геномики и мобиломики растений МФТИ.
Но процесс поиска разных аллелей осложняется тем, что большинство генов сельскохозяйственных растений достаточно длинные (3000–5000 пар нуклеотидов). Как известно, последовательность генов обозначается буквами. Код ДНК состоит из четырех нуклеотидов: А (аденин), Т (тимин), Г (гуанин) и Ц (цитозин). При этом для определения наилучших качеств сорта недостаточно прочесть только первый десяток букв. Варианты могут отличаться либо на одну, либо на десятки букв, которые распределены по всей весьма внушительной длине.
Источник: Agronomy
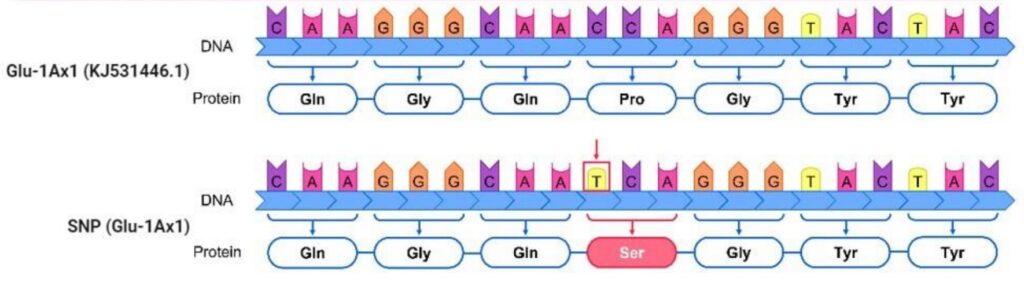
«Распознание аллелей генов может занять много времени. Например, мы берем растение с последовательностью в несколько тысяч букв, при этом у одного аллеля этого гена в 10-м положении одна буква, у другого — в 20-м, и так далее. В итоге для четкого понимания будущего качества нам необходимо расшифровать все последовательности, а это весьма дорого. Раньше использовали весьма экономный метод белкового электрофореза, который позволял разделить состав зерна на индивидуальные белки и просчитать до десятка вариантов генов. Но сейчас мы знаем, что этих вариантов больше сотни. В итоге ученые решили найти более эффективный метод и предложили отбор вариантов по фиксированному положению нуклеотидов. Например, у одного варианта глютена нуклеотид аденин в этом положении, у другого в этом положении уже гуанин, а у третьего — тимидин. Это кажется лучше, но мы же помним, что у нас сотни вариантов и эти замены распределены по всему гену. В идеале необходимо расшифровать весь ген и сделать это не у десятков, а у сотни образцов. В итоге эта важная и актуальная ниша осталась незаполненной», — подчеркнул Илья Киров.
В ходе исследования к решению этой задачи ученые решили подойти альтернативным, но хорошо известным им путем: секвенированием с возможностью делать длинные прочтения (риды). Этот вид секвенирования не ограничен по размеру ДНК. Таким образом, можно расшифровать и последовательности в сотни тысяч нуклеотидов. Подобным методом уже успешно расшифровали гены бактерий и человека, но на растениях данный метод особо не применялся.
«Секвенирование длинными ридами намного быстрее и дешевле классического метода секвенирования по Сэнгеру. Мы решили опробовать этот метод на генах глютенинов с протяженностью гена около 4000 нуклеотидов и большим числом повторяющихся последовательностей. В итоге за несколько дней мы расшифровали шесть полноразмерных генов глютенинов 23 сортов пшеницы. При этом само секвенирование занимает два часа, и за это время мы расшифровали 120 последовательностей фрагментов ДНК по 4000 нуклеотидов. Нам удалось не только точно определить где в гене, какие и в каком положении произошли замены, кодирующие белок, но и определить изменения последовательности, которая контролирует наработку РНК. Это уже промоторная область, играющая ключевую роль в процессе инициации транскрипции — передачи наследственной информации с молекулы ДНК на РНК. Замену в этой области белковый электрофорез не показывает. К тому же новый подход позволил нам найти новый вариант гена глютенина, который еще предстоит изучить», — подытожил Илья Киров.
Таким образом, ученые подобрали и оптимизировали более экономичную систему расшифровки отдельных генов растений для молекулярной селекции. Еще один плюс данного метода в том, что его результаты не противоречат классическому белковому форезу, который считается золотым стандартом, но при этом не находит варианты, которые видит метод секвенирования длинными ридами. Это очень важно, так как замена даже одной буквы (аминокислоты) в последовательности белка может изменить его поведение, нарушить физико-химические свойства и в итоге привести к другому фенотипу.
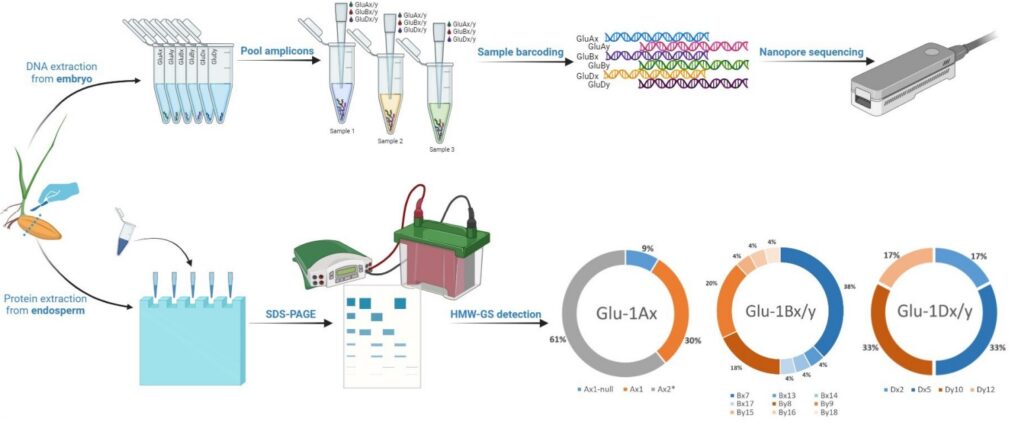
Схема, изображающая идентификацию генов глютенинов. Каждую зерновку делили на две части: эндосперм использовали для традиционных методов генотипирования SDS-PAGE, а проростки, полученные из зародыша, подвергали выделению ДНК и расшифровке целевых генов глютенинов. Источник: Agronomy

